近日,中国水产科学研究院黄海水产研究所海洋渔业资源分子生态学研究团队成功破译了鳀(Engraulis japonicus)染色体水平基因组,成果以“Chromosome-level genome assembly and annotation of Japanese anchovy (Engraulis japonicus)”为题发表在Scientific Data上。这是该团队在海水鱼类资源分子生态研究方向取得的又一重要成果,是基因组学技术在渔业资源生态研究的拓展应用,为深入理解鱼类资源对环境适应的分子机制和管理海洋鱼类种群提供了科学依据。

鳀是西北太平洋重要的中上层小型鱼类,主要分布在我国渤、黄、东海,朝鲜半岛沿海以及日本周边海域。鳀曾是我国黄东海单一物种资源量最大的鱼种,20世纪90年代年渔获量高达百万吨;鳀也是黄东海食物网关键种,其资源兴衰直接影响到蓝点马鲛、带鱼等肉食性经济鱼类的饵料供应,在渔业生态系统食物链中承担能量传递的“桥梁”作用,具有极高的生态价值和经济价值。进入新世纪以来,黄东海鳀资源急剧衰退,渔获物低龄化、小型化现象明显,群体密度日益下降,目前已难以形成渔汛。不言而喻,鳀资源的可持续利用不仅关系区域渔业经济,更是维护黄东海生态系统稳定的关键所在。该团队所发布的高质量鳀基因组为进一步挖掘该物种的遗传资源和研究小型中上层鱼类适应性进化以及资源高值化技术奠定数据基础。
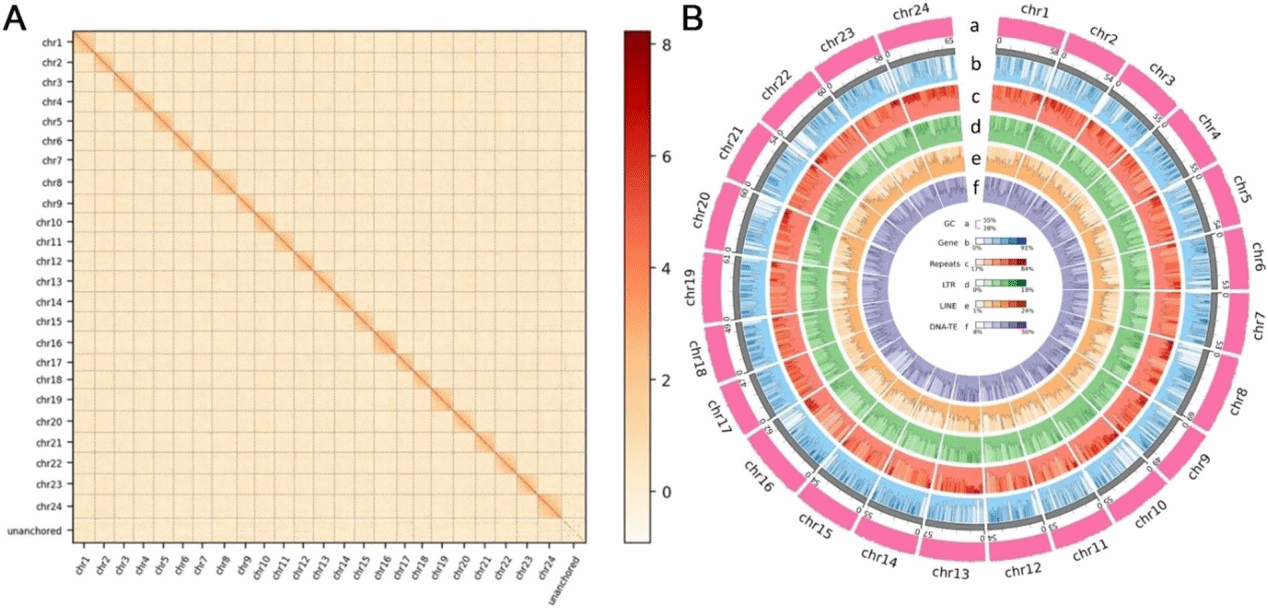
研究团队采用PacBio HiFi、Hi-C技术和Illumina短读测序技术,首次构建了鳀的高质量染色体水平基因组。该基因组大小为1467.6Mb,contig N50长度为456.3kb,其中1423.3MB锚定至24条染色体,覆盖95.2%的组装序列。BUSCO评估显示基因组完整性高达94.07%;注释了780.9Mb(54.9%)重复序列,重复序列中,长散在核元件(LINE)、短散在核元件(SINE)、长末端重复序列(LTR)和卫星DNA序列分别占基因组的6.3%、0.9%、9.1%和2.7%;组装基因组杂合度高达2.3%;预测了24,405个蛋白编码基因,其中97.15%的功能基因获得数据库注释,同时预测出23984个非编码RNA(ncRNAs);经组装准确性和连续性验证,HiFi和Hi-C数据映射率达100%,Merqury QV值为49.74。
论文链接:https://doi.org/10.1038/s41597-025-04423-z