极地海洋与深海作为地球上复杂且特殊的海洋生态系统,常年面临着低温、高扰动等极端环境的严峻挑战,但却孕育出复杂而多样的海洋生物,展现了生命体在地球极端条件下的显著韧性和多样性。海洋生物的极端环境适应性是一个复杂的生物学问题,涉及基因组结构及表达特征的变化、群体遗传多样性的重塑、微生物共生关系的建立等多个方面,进而引发海洋生物生理、行为改变的一系列生态适应和进化过程。
南极磷虾是极地南大洋生态系统中资源量最丰富的奠基性物种,深海珊瑚则是深海生态系统形成的关键工程物种。依托国家海洋水产种质资源库,中国水产科学研究院黄海水产研究所邵长伟研究员团队以南极磷虾和深海珊瑚为研究对象,综合运用基因组、宏基因组等多组学技术,发掘了极地和深海生物海量基因资源,进一步从宿主基因组进化、线粒体适应性到宿主-共生菌协同互作等多个维度,成功揭示了极端环境生物适应的通用法则与独特策略。上述工作将国家库保藏的海洋生物基因资源发掘范围由近海、远洋拓展到极地海洋与深海,开辟了海洋生物基因资源高效开发与利用的新途径。相关成果发表于《Cell》(封面论文)、《Cell Host & Microbe》《Zoological Research》(封面论文)、《mSystems》《BMC genomics》等国际知名期刊。
面对南极剧烈的季节变化,南极磷虾演化出了极大的基因组(48.01Gb)。研究揭示,其基因组庞大体量的核心在于基因间转座元件的爆发性扩张。更为重要的是,研究首次鉴定并解析了磷虾完整的生物钟通路,为实现与极端光周期的高度同步奠定了分子基础。种群基因组分析进一步表明,尽管磷虾群体在环南极尺度上呈现高度均质化,但微弱的环境选择信号正塑造着其遗传结构,历史动态更与百万年来的全球气候变化事件相耦合(Cell,2023)。此外,通过南极磷虾80个个体线粒体的群体基因组学分析,发现南极磷虾线粒体具有极高的遗传多样性,并经历了近期的种群扩张。线粒体蛋白编码基因均受到强烈的纯化选择,表明其核心能量代谢功能在极端环境下高度保守(BMC Genomics,2025)。
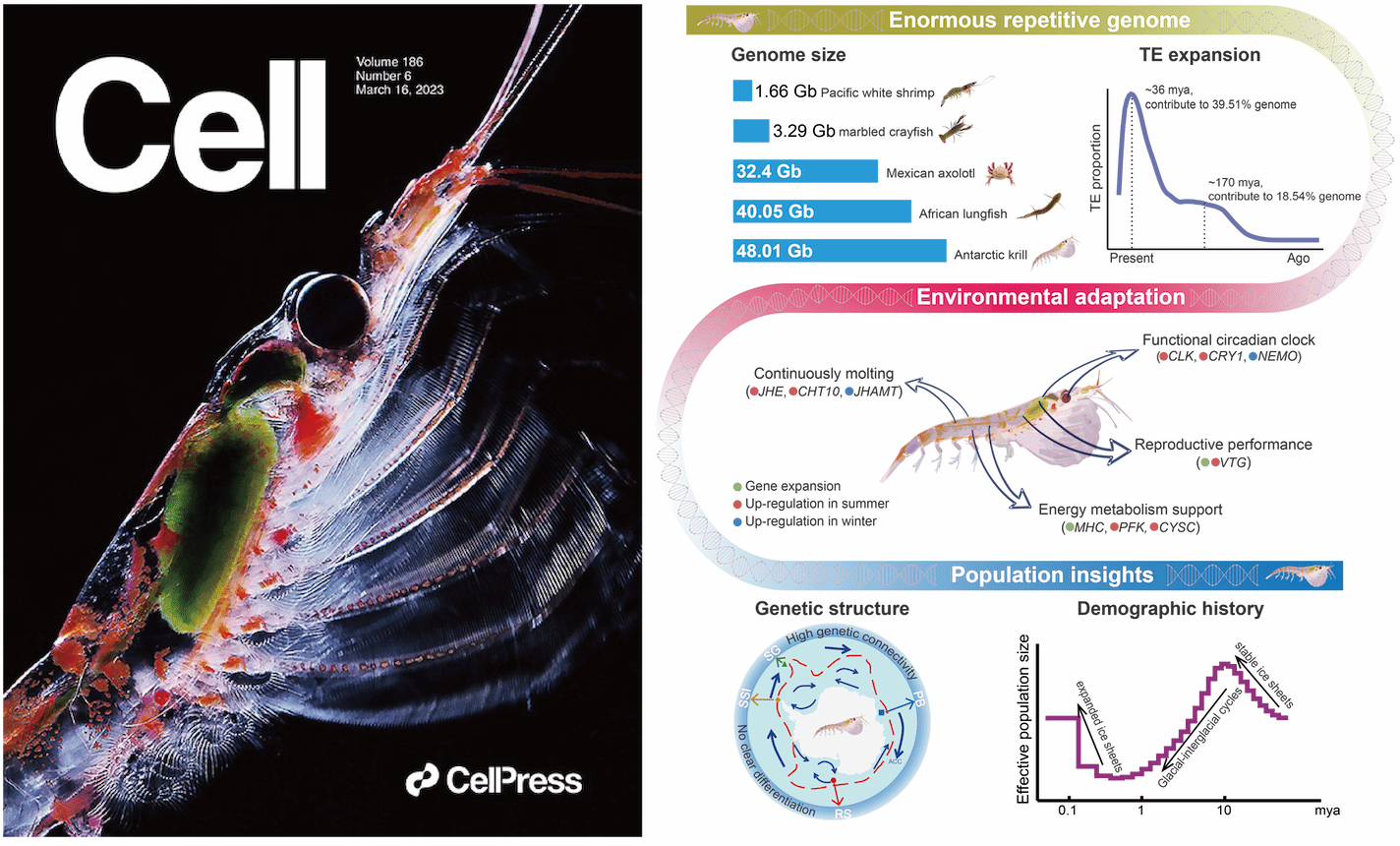
南极磷虾基因组变化揭示了对南极环境适应的机制
除了自身基因组创新,南极磷虾还通过与肠道共生菌建立高效的协同关系适应极端的环境。团队首次系统解析了南极磷虾肠道菌群的功能,鉴定出12个高丰度的新菌群,根据代谢潜能将它们分为两大功能簇,分别参与宿主降解有机物过程和合成宿主所需多种氨基酸、维生素。转录组数据证实肠道共生菌代谢活跃,它们如同一个内置的、可动态调控的营养补给站和抗氧化剂合成工厂,极大地增强了南极磷虾在营养波动和氧化应激环境中的生存韧性(mSystems,2025)。
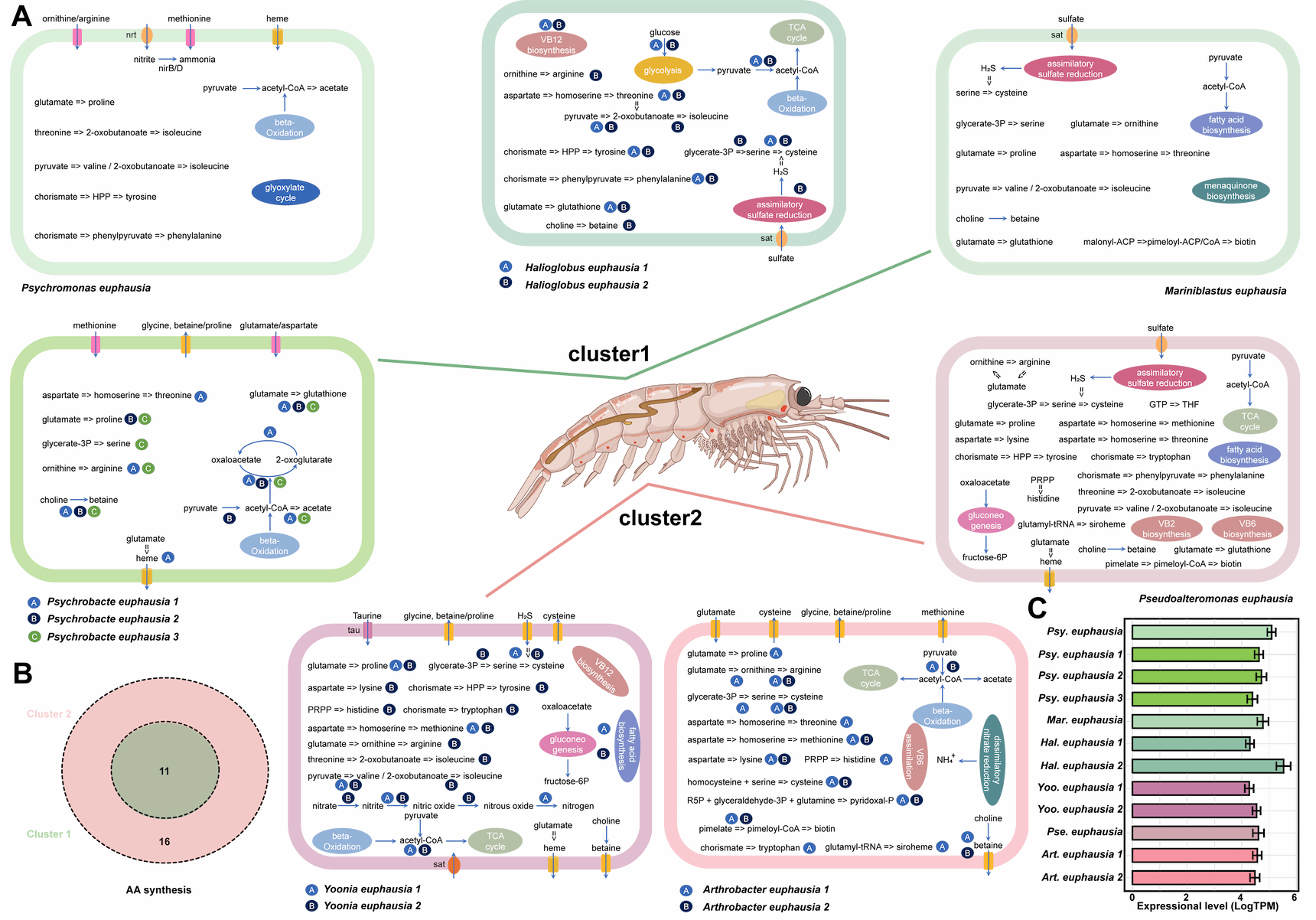
南极磷虾肠道微生物群的代谢模式图
在深邃的黑暗世界中,深海珊瑚则展示了另一套生存哲学。研究团队对来自南海的八方珊瑚与六方珊瑚研究发现,深海珊瑚的线粒体进化速率极为缓慢。线粒体基因排列顺序高度保守,13个蛋白编码基因均受纯化选择主导。这种以不变应万变的进化策略,可能与深海环境长期稳定及其体内特有的DNA修复蛋白有关,使得它们的线粒体成为记录深部演化历史的分子活化石(Zoological Research,2024;International Journal of Molecular Sciences,2024)。然而,仅凭线粒体难以窥见全貌。研究团队进一步绘制了伪交替深海黑珊瑚染色体水平的基因组图谱,发现转座元件的近期爆发与有效种群大小的峰值同步,可能驱动了基因组的重塑与适应。深海黑珊瑚在营养转运、免疫识别和溶酶体消化等相关基因家族的显著扩张,为它容纳并管理共生菌群奠定了坚实的遗传平台(Cell Host & Microbe,2025)。
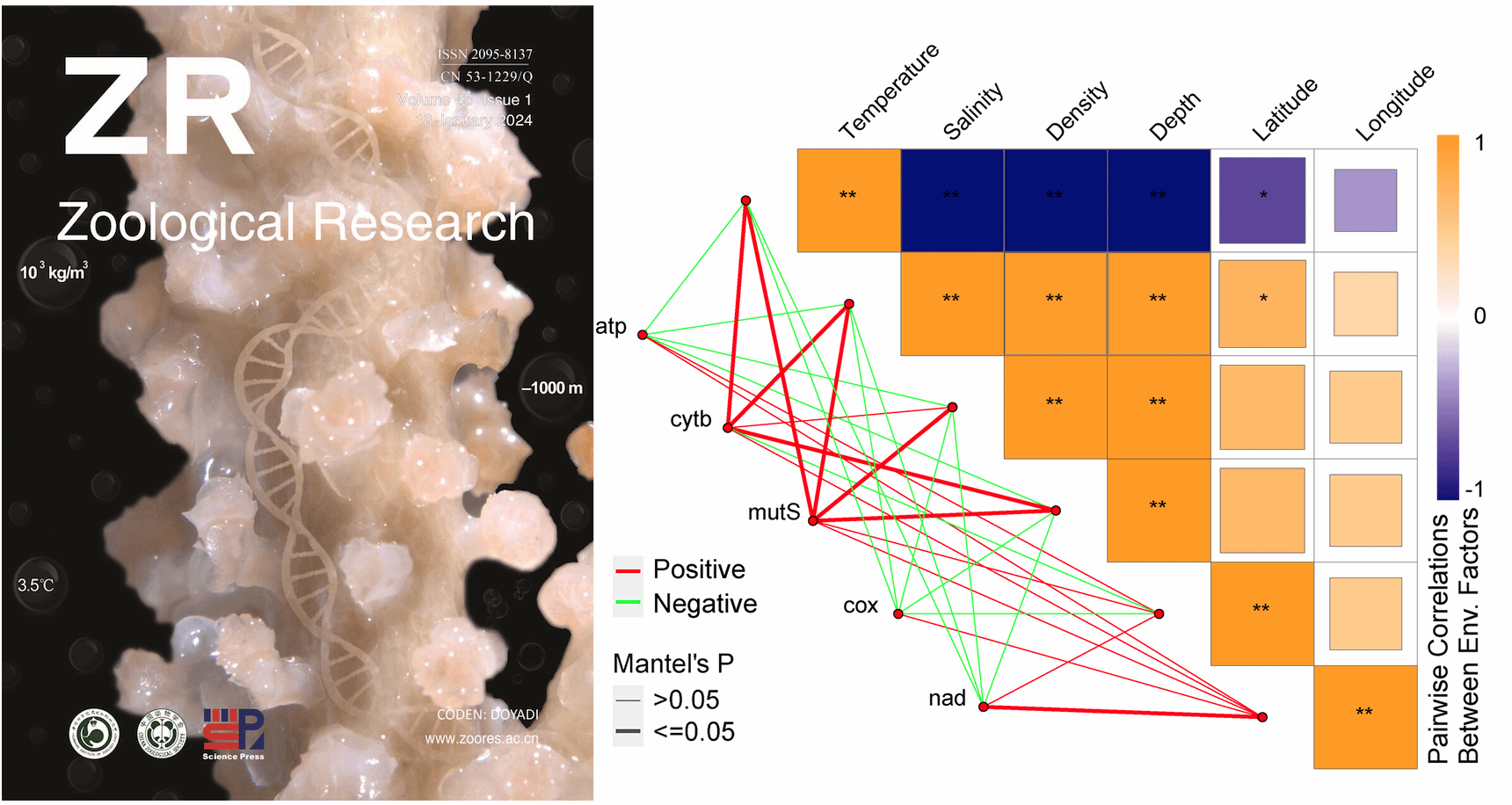
珊瑚线粒体基因 Ka/Ks与环境变量之间的 Mantel 检验
与磷虾肠道菌群的高多样性不同,深海黑珊瑚的共生菌群呈现出精简而稳定的特征。通过多地点、多深度样本证实其体内主要由三大类共生菌主导,主要集中于水螅体的中胶层,形成一个高度特化的内部微生态系统。这种简化模式暗示着宿主通过长期协同进化,主动筛选并维系了一个功能互补、效率极高的核心共生群体。此外,研究提出了宿主-共生菌稳态调控分子模型,即宿主通过免疫反应、吞噬与溶酶体消化等机制,有效维持与共生菌的平衡,实现长期稳定共存(Cell Host & Microbe,2025)。
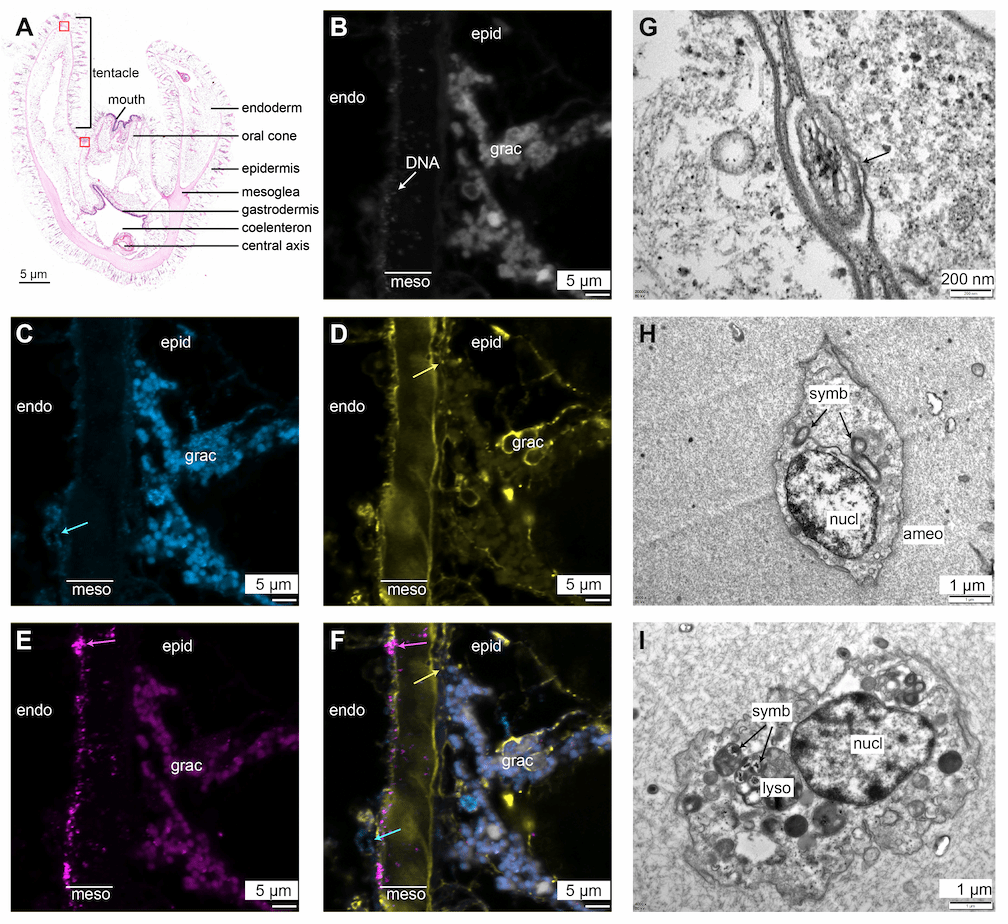
共生菌在深海珊瑚中胶层中的定位
综上,作为地球上重要的两大海洋生态系统,虽远隔重洋,却揭示了极端环境下共同的生存智慧:一方面,生物通过自身基因组的可塑性构建适应性根基;另一方面,它们均发展与微生物的共生联盟实现个体能力的超越。这些研究从遗传基础和生态策略层面阐明了关键物种适应并维系极端生态系统的内在机制,为评估未来环境变化对海洋生物资源的影响提供了理论基础,也为海洋生物基因资源的可持续开发利用提供了关键的技术路径。
文章链接:
Shao, C., Sun, S., Liu, K., Wang, J., Li, S., Liu, Q., Deagle, B., Seim, I., Biscontin, A., Wang, Q., et al. (2023). The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights. Cell 186, 1279-1294. 10.1016/j.cell.2023.02.005.
Sun, S., Li, S., Seim, I., Du, X., Yang, X., Liu, K., Wei, Z., Shao, C., Fan, G., and Liu, X. (2025). Complete mitogenomes reveal high diversity and recent population dynamics in Antarctic krill. BMC Genomics 26, 419. 10.1186/s12864-025-11579-w.
Wei, Z., Meng, L., Du, S., Wang, F., Jiang, A., Lu, R., Liu, K., Wang, X., Xu, Q., Mu, X., et al. (2025). Metabolic potentials of the gut microbes in Antarctic krill (Euphausia superba). mSystems 10, e00377-00325. 10.1128/msystems.00377-25
Wei, Z., Lan, Y., Meng, L., Wang, H., Li, L., Li, Y., Zhang N., Lu, R., Cui, Z., Song, Y., et al. (2025). Hologenomic Insights into the Molecular Adaptation of Deep-sea Coral Bathypathes pseudoalternata. Cell Host & Microbe. 10.1016/j.chom.2025.10.020
Wei, Z., Ta, K., Zhang N., Liu S., Meng L., Cai C., Peng X., and Shao C. (2024). Molecular phylogenetic relationship based on mitochondrial genomes within novel deep-sea corals (Octocorallia: Alcyonacea) insights into the slow evolution and adaptation of the extreme deep-sea environment. Zoological Research. 10.24272/j.issn.2095-8137.2023.039
Wei, Z., Yang, Y., Meng, L., Zhang, N., Liu, K., Meng, L., Li, Y., and Shao, C. (2024). The Mitogenomic Landscape of Hexacorallia Corals: Insight into Their Slow Evolution. International Journal of Molecular Sciences 25, 8218. 10.3390/ijms25158218.

